====== Apprentissage automatique pour l'exploration d'imagerie moléculaire par spectrométrie de masse ======
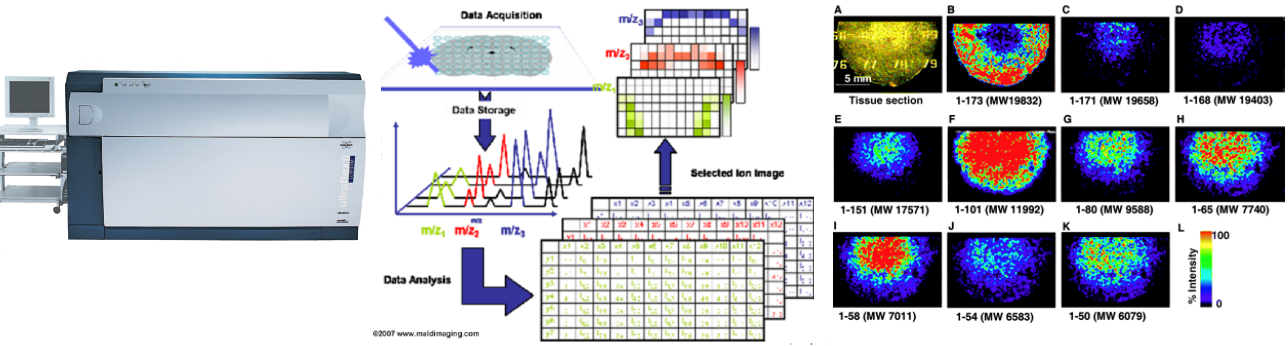
Le but de ce travail est d'évaluer des méthodes d'apprentissage
automatique supervisé (p.ex.: arbres de décision, SVMs, ...) et non-supervisé (p.ex. clustering hiérarchique, ...) pour faciliter l'exploration de
données d'imagerie moléculaire par spectrométrie de masse.
L'étudiant travaillera en collaboration avec notre équipe et le [[http://www.giga.ulg.ac.be/jcms/prod_183306/laboratoire-de-spectrometrie-de-masse|Laboratoire de Spectrométrie de Masse]] du GIGA
sur des images d'histologie moléculaire relative au cancer.
====== Bibliographie ======
* [[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2522327/pdf/418_2008_Article_469.pdf|MALDI imaging mass spectrometry for direct tissue analysis: a new frontier for molecular histology, 2008]]
* [[http://pubs.acs.org/doi/pdf/10.1021/pr8005777|MALDI Imaging Combined with Hierarchical Clustering as a New Tool for the Interpretation of Complex Human Cancers, 2008]]
* [[http://www.montefiore.ulg.ac.be/services/stochastic/pubs/2009/MSGGM09/|Marée et al. A Machine Learning Approach for Material Detection in Hyperspectral Images, 2009]]
====== Renseignements ======
[[http://www.montefiore.ulg.ac.be/~maree/|Raphaël Marée]] () et Prof. Edwin De Pauw ()
